共找到18條詞條名為王艷麗的結果 展開
王艷麗
中國科學院生物物理研究所研究員
王艷麗,女,畢業於中國科學技術大學,中科院生物物理所研究員,博士生導師。中科院“百人計劃”獲得者,非編碼核酸北京市重點實驗室創新課題組組長。
主要研究方向為RNA干擾(RNA interference,RNA)相關蛋白的結構與功能研究。
| 時間 | 院校 | 學位 |
| 2002年—2004年 | 中國科學技術大學 | 博士 |

王艷麗老師
2006年—2011年擔任美國斯隆-凱瑟琳癌症研究中心博士后、副研究員、高級研究員。
2011年—至今擔任中國科學院生物物理研究所“百人計劃”研究員。
研究方向
目前王艷麗研究員課題組的研究工作主要集中在以下兩個方面。
(1)CRISPR/Cas系統的作用機理研究
CRISPR/Cas系統是源於細菌和古細菌的一種後天免疫系統,它是近年來發現的由小分子RNA介導的免疫系統。CRISPR/Cas廣泛存在於原核生物中的一種由crRNA介導干擾系統,分為2大類。每一類包含不同的類型,其中,1類分為I型、III型、IV型;2類分為II型、V型、VI型。Ⅰ型CRISPR系統的crRNA加工成熟后,與多個Cas蛋白結合形成cascade複合物,通過crRNA序列特異性地識別,並招募Cas3蛋白對外源入侵核酸序列剪切。
本課題組成功解析了解析度為3埃的E.coli Cascade複合物結構,揭示了由11個Cas蛋白以及一個61核苷酸的crRNA共同組成的分子量為405kDa的Cascade複合物的精確的組裝方式,揭示了第一類中Ⅰ型CRISPR系統抗病毒的分子機理,同時也為進一步了解靶標的識別機制提供了新的信息(Nature, 2014)。發現了Cas1-Cas2識別外源入侵DNA分子機制,揭示了外源核酸片段的長度是如何確定的。該成果為揭示原核生物這一新的抵禦病毒及遺傳物質的入侵的機制奠定了重要的理論基礎 (Cell, 2015)。通過解析AcrF3-Cas3複合物結構,闡明了anti-CRISPR 蛋白AcrF3對抗Ⅰ型CRISPR/Cas系統的作用機制,對病毒與宿主共同進化在分子層面提供了新的見解(Cell Res.2016)。
還對第二類CRISPR進行了研究。解析了結合有sgRNA的C2c1晶體結構,發現目的序列的鹼基突變顯著降低C2c1切割活性,該研究結果有助於開發新的基因組編輯工具,降低基因編輯過程中的脫靶現象(Mol Cell,2017);還解析了C2c2和C2c2-crRNA的晶體結構,發現C2c2結構不同於其它Cas蛋白,揭示了C2c2剪切pre-crRNA以及切割靶標RNA的分子機制,對理解細菌抵抗RNA病毒入侵的分子基礎具有十分重要的意義;同時也為改造CRISPR-C2c2系統在基因編輯領域的運用提供了強有力的理論依據(Cell,2017)。
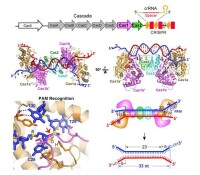
DNA複合物的晶體結構
在小分子RNA介導的基因沉默的過程中,探索Ago蛋白與嚮導鏈的構象變化,深入分析miRNA誘導基因沉默的機理。RNAi是指在進化過程中高度保守的、由雙鏈RNA介導的特異性基因沉默現象。Argonaute(Ago)蛋白是RNA基因沉默途徑中核心元件,其PIWI結構域能夠催化引導鏈介導的mRNA的特異性降解。通過解析argonaute蛋白與引導鏈及靶RNA等複合物的結構,詳細解釋了argonaute沉默複合體參與調控基因沉默的具體途徑和方式,闡明了其如何選擇並切割RNA分子,最終降解RNA分子使基因沉默的過程。
發現某些原核生物的Ago蛋白不僅具有切割RNA的功能,還具有在小DNA分子介導下序列特異性的切割DNA的功能。通過解析系列的Ago蛋白與嚮導DNA以及靶DNA的三元複合物結構,揭示了Ago蛋白切割DNA的分子機理(PNAS,2014)。
代表性論文
1.Liu L, Li X, Wang J, Yin M, Chen P, Wang M, Li J, Sheng G, Wang Y. Two Distant Catalytic Sites Are Responsible for C2c2 RNase Activities. Cell. 2017,168:121-134.
2.Liu L, Chen P, Wang M, Li X, Wang J, Yin M, Wang Y. C2c1-sgRNA Complex Structure Reveals RNA-guided DNA Cleavage Mechanism. Molecular Cell. 2017, 65.
3.Wang J, Ma J, Cheng Z, Meng X, You L, Wang M, Zhang X, Wang, Y. A CRISPR evolutionary arms race: structural insights into viral anti-CRISPR/Cas responses. Cell Research. 2016, 26:1165–1168.
4.Wang, J., Li, J., Zhao, H., Sheng, G., Wang, M., Yin, M. & Wang, Y., Structural and Mechanistic Basis of PAM-Dependent Spacer Acquisition in CRISPR-Cas System. Cell. 2015, 163: 840-853.
5.Zhao, H., Sheng, G., Wang, J., Wang, M., Bunkoczi, G., Gong, W., Wei, Z. & Wang, Y. “Crystal structure of the RNA-guided immune surveillance Cascade complex in Escherichia coli”, Nature. 2014, 151: 147-150.
6.Swarts, D. C., Makarova, K., Wang, Y., Nakanishi, K., Ketting, R., Koonin, E., Patel, D. J. & Oost, van der, J., “The evolutionary journey of Argonaute proteins”, Nature structural & molecular biology, 2014, 21: 743-753.
7.Swarts, D. C., Jore, M. M., Westra, E. R., Zhu, Y., Janssen, J. H., Snijders, A. P., Wang, Y., Patel, D. J., Berenguer, J., Brouns, S. J.J. & Oost, van der, J. "DNA-guided DNA interference by a prokaryotic Argonaute", Nature. 2014, 507: 258-261.
8.Sheng, G., Zhao, H., Wang, J., Rao, Y., Tian, W., Swarts, D. C., van der Oost, J., Patel, D. J. and Wang, Y. "Structure-based cleavage mechanism of Thermus thermophilus Argonaute DNA guide strand-mediated DNA target cleavage." Proc Natl Acad Sci U S A.2014, 111(2): 652-657.
9.Swarts, D. C., Jore,M. M., Westra,E. R., Zhu, Y., Janssen, J. H., Snijders,A. P., Wang, Y., Patel, D. J., Berenguer, J., Brouns, S. J. and van der Oost, J. (2014). "DNA-guided DNA interference by a prokaryotic Argonaute." Nature 507(7491): 258-261.
10.Rüdel S, Wang, Y, Lenobel R, K?rner R, Hsiao HH, Urlaub H, Patel D, Meister G. Phosphorylation of human Argonaute proteins affects small RNA binding. Nucleic Acids Res. 2011,39:2330-43.
11.Wang, Y., Ludwig J., Schuberth C., Goldeck M., Schlee M., Li H., Juranek S., Sheng G., Micura R., Tuschl T., Hartmann G., Patel D., Structural and functional insights into 5'-ppp RNA pattern recognition by the innate immune receptor RIG-I. Nat Struct Mol Biol. 2010, 17:781-7.. 2010, 17:781-7.
12.Wang, Y, Juranek S, Li H, Sheng G, Tuschl T, Patel DJ. Nucleation, propagation and cleavage of target RNAs in Ago silencing complexes. Nature. 2009, 461:754-761.
13.Wang, Y, Juranek S, Li H, Sheng G, Tuschl T, Patel DJ. Structure of an argonaute silencing complex with a seed-containing guide DNA and target RNA duplex. Nature. 2008, 56:921-926.
14.Wang, Y, Sheng G, Juranek S, Tuschl T, Patel DJ., Structure of the guide-strand-containing argonaute silencing complex. Nature. 2008, 456:209-213.
15.Wang, Y, Liu L, Wei Z, Cheng Z, Lin Y, Gong W. Seeing the process of histidine phosphorylation in human bisphosphoglycerate mutase. J Biol Chem. 2006, 281:39642-8.
16.Wang, Y, Wei Z, Liu L, Cheng Z, Lin Y, Ji F, Gong W. Crystal structure of human B type phosphoglycerate mutase bound with citrate. Biochem. Biophys. Res. Commun. 2005, 331:1207-15.
17.Wang, Y, Wei Z, Bian Q, Cheng Z, Wan M, Liu L, Gong W,Crystal structure of human bisphosphoglycerate mutase,J. Biol. Chem. 2004, 279: 39132-8.
| 時間 | 獎項全稱 |
| 2017年02月 | 第九屆“談家楨生命科學創新獎” |
| 2016年11月 | 第十三屆“中國青年女科學家獎” |
