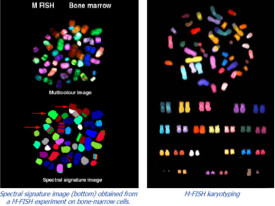
共找到7條詞條名為fish的結果 展開
fish
熒光原位雜交技術
熒光原位雜交技術(fluorescence in situ hybridization),簡稱FISH。是利用熒游標記的特異核酸探針與細胞內相應的靶DNA分子或RNA分子雜交,通過在熒光顯微鏡或共聚焦激光掃描儀下觀察熒光信號,來確定與特異探針雜交后被染色的細胞或細胞器的形態和分佈,或者是結合了熒光探針的DNA區域或RNA分子在染色體或其他細胞器中的定位。
1969年,Pardue和John等兩個研究小組開始採用放射性標記DNA或28S RNA發明了原位雜交技術(ISH)。儘管當時原位雜交技術已經具有較高的特異性和靈敏度,但鑒於放射性同位素自身特性的局限,如安全性、空間解析度低、不穩定性等問題,這項技術僅限於實驗室研究方面的應用。
1986年科研工作者開始利用異硫氰酸鹽熒光素來標記探針,並在熒光顯微鏡下進行觀察分析,建立了熒光原位雜交技術(FISH)。
1989年,Delong首次使用熒游標記寡核苷酸探針檢測單個微生物細胞。由於FISH技術具有敏感度高、信號強、背景低、快速等優點,該方法在環境微生物的檢測中得到了廣泛的應用。
隨著科技的迅速發展,FISH探針標記物越來越多,不僅從單一熒光發展到多色熒光檢測,而且應用範圍也進一步擴大,不僅可以用於分裂相細胞而且可以用於間期細胞檢測,為FISH技術的臨床應用打下了堅實的基礎。
(1)樣品的固定;
(2)樣品的製備和預處理;
(3)預雜交;
(4)探針和樣品變性;
(5)用不同的探針雜交以檢測不同的靶序列;
(6)漂洗去除未結合的探針;
(7)檢測雜交信號,進行結果分析
·熒光信號觀察:
將處理好的樣品置於熒光顯微鏡下,選擇分散較好的區域來觀察。三色(或者更多)熒光激發下,觀察到不同顏色的熒光圖像。通常選用20X物鏡來掃描樣品雜交區域,40X或100X物鏡下觀察樣品,從一定的方向進行計數,並對計數情況進行分析。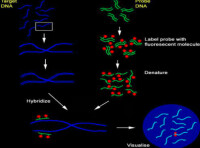

fish操作步驟
·FISH最常使用的靶序列是16S rRNA,根據rRNA目標區域可設計寡核苷酸探針,進行種屬特異性鑒定;
(1)寡核苷酸探針FISH技術
·最常用的寡核苷酸探針一般是15-30 bp,短的探針易於結合到靶序列,但一般很難被標記;·探針的標記有直接標記和間接標記兩種;
(2)核酸肽探針FISH技術
·核酸肽(Peptide Nucleic Acid,PNA):一種不帶電荷的DNA類似物,其主鏈骨架是由重複的N-(2-氨基乙基)甘氨酸以醯胺鍵聚合而成,鹼基通過亞甲基連接到PNA分子的主鏈上。·PNA/DNA分子雜交結合力、專一性都很高,由於沒有電荷排斥力,使其雜交形成雙螺旋結構的熱穩定性高;·PNA探針能夠接近位於rRNA高級結構域中的特異靶序列,極大地提高了PNA-FISH檢測的靈敏性;·PNA探針一般長度為15 bp;
(3)多彩(Multicolor)FISH技術
·近年來,很多學者致力於以不同染色的標記探針和熒光染料同時檢測出多個靶序列的研究,最新的多彩FISH技術可以同時用七種染色進行檢測;
·應用多彩FISH時,應注意幾個問題:
A. 採用多波峰的濾鏡;
B. 混合的熒光染料應該具有狹窄的散射峰,以防止探針間光譜重疊,從而去除背景和避免褪色等問題;
C. 在檢測低丰度靶序列時,應採用光穩定的高亮度染料;
(1)監測微生物群落結構與群落動態
·FISH技術多用於分析單個細胞水平上的微生物群落結構,以rDNA為靶點的寡核苷酸探針,可用於原位鑒定單個細胞;
·以16S rRNA為靶序列的FISH技術,可用於檢測活性污泥微生物群落的種群組成和數量水平。同時,可以對特異菌群進行空間定位和原位生理學分析;
如:在高濃度氨工業廢水中,通過16S rRNA為靶序列的FISH技術證實硝化桿菌類細菌存在於活性污泥中;並且,硝化球菌是優勢種群。
·目前,在歐美等國,rRNA測序的群落分析已經廣泛應用於監測生物反應器或廢水處理廠的生物相;·應用共聚焦激光掃描顯微鏡(CLSM)的FISH技術可以對生物膜和活性污泥絮體的特異種群進行空間分佈研究;
(2)FISH技術結合流式細胞儀檢測微生物
·是診斷和評價微生物群落結構及其動態的最有前景的技術,是近幾年來分析土壤活性污泥等環境樣品中微生物分揀和數量的有效手段,適用於對有關微生物群落進行快速和頻繁的監測,而且自動化操作水平高;
(3)FISH技術應用時的主要問題
·FISH檢測的假陽性可能原因:(a)探針設計不合理;(2)微生物自身熒光的干擾;(c)環境樣品(如活性污泥或飲用水等)中天然的可發熒光的生物或化學殘留物的干擾;(d)培養基、固定方法和封固劑等對熒光信號強度的影響;
·FISH檢測的假陰性
可能原因:(a)細胞壁的結構影響探針的滲透力,導致雜交信號強度降低。革蘭氏陽性菌必須進行特殊的固定和前處理,以提高探針的滲透力;(b)細菌細胞中rRNA含量過低;