RNA-seq
轉錄組測序技術
RNA-seq測序和分析
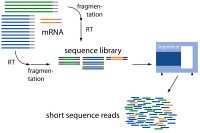
RNA測序最經常用於分析差異表達基因(DEG)。標準的工作流程從實驗室提取RNA開始,到mRNA富集或去除核糖體RNA,cDNA反轉錄以及製備由接頭連接的測序文庫。接下來,這個文庫會被高通量測序平台測序,每一個樣本通常會被測序到10000000~30000000讀長。最後,實驗得到的數據通過比對或拼接測序的讀長到轉錄組,量化覆蓋轉錄本的讀長,過濾和樣本間歸一化,用統計模型描述每個基因在各個樣本組之間存在什麼樣的表達水平上的差異。
早期RNA-Seq實驗使用組織測序分析差異基因,在不同的生物中都可以分析,比如玉米,擬南,釀酒酵母,小鼠,人類等。RNA測序可以被當作不同方法或者生物學應用的總稱,其中差異基因的分析始終是RNA測序的主要應用場景,被當做是一個常規的實驗手段。
RNA測序更廣泛的應用場景加深了我們對於很多生物學領域的理解,比如mRNA剪切程度,non-coding RNA以及enhancer RNA對於基因表達的調節。RNA測序的發展被濕實驗和計算方法兩者的進步共同驅動,帶來了RNA生物學豐富而又更加客觀地認識,也讓轉錄組學的發展在之前基因晶元技術的基礎上成為可能。截止2019年,存在眾多不同的RNA測序流程,大多數由Illumina提供,但也有長讀長RNA測序以及直接RNA測序(dRNA-seq)的技術進步解決了Illumina為代表的短讀長測序技術所不能解決的問題。
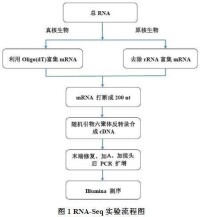
樣品提取總RNA后,對於真核生物,用帶有Oligo(dT)的磁珠富集mRNA,對於原核生物,用試劑盒去除rRNA,向得到的mRNA中加入Fragmentation Buffer使其片斷成為短片段,再以片斷後的mRNA為模板,用六鹼基隨機引物(random hexamers)合成cDNA第一鏈,並加入緩衝液、dNTPs、RNase H 和DNA polymerase I 合成cDNA第二鏈,經過QiaQuick PCR試劑盒純化並加 EB緩衝液洗脫經末端修復、加鹼基A,加測序接頭,再經瓊脂糖凝膠電泳回收目的大小片段,並進行PCR擴增,從而完成整個文庫製備工作,構建好的文庫用Illumina HiSeq2000進行測序。
RNA-seq
高靈敏度:能夠檢測到細胞中少至幾個拷貝的稀有轉錄本。
全基因組分析:可以對任何物種進行全基因組分析。無需預先設計特異性探針,因此無需了解物種基因信息,能夠直接對任何物種進行轉錄組分析。同時能夠檢測未知基因,發現新的轉錄本,並精確地識別可變剪切位點及cSNP,UTR區域。
檢測範圍:高於6個數量級的動態檢測範圍,能夠同時鑒定和定量稀有轉錄本和正常轉錄本。